FAQ
GlyfinTMS についての質問とその回答です。
【FAQ 全般】【FAQ 動作全般】
- ReAdWを指定フォルダに配置してもデータが読み込めません。 →回答
- ファイルがオープンできません →回答
- うまく検出してくれません →回答
- 解析パラメータ調節を効率良く行ないたい →回答
- 「抽出」と「検出」の違いは? →回答
- 解析結果を保存したい →回答
【FAQ パラメータ関連】
GlyfinTMS についての質問とその回答です。
【FAQ 全般】
(グライフィンティエムエス: Glyco peak Finder from Tandem Mass Spectrometry)の略と、JAMSTEC発のソフトウェアで世界の糖鎖(Glyco)研究を飛躍推進させたいと言う意味合いで、魚の鰭(Fin)=翼の造語として考案された。
商標取得 :「GlyfinTMS」 (文字商標) 第 9 類、 42 類 登録番号 第 5319330 号
申し訳ありません。
現在の GlyfinTMS は、Windows OS のみに対応しています。
対応OS は以下のとおりです。
・Windows XP
・Windows Vista
・Windows 7
【Q】
ReAdW(v4.3.1)を指定されたフォルダに配置していますが、データを読み込めません。
【A】
下記の図のように、ReAdWの実行ファイルとzlib1.dllは「c:\Xcalibur\data\」の下に置いてください。
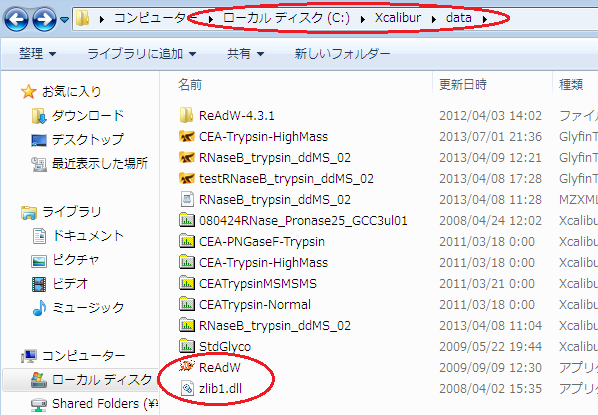
【Q】
ファイルがオープンできません。
【A】
GlyfinTMS では、以下の入力データに対応しています。
解析可能な raw data ファイルフォーマットと、対応する分析器は以下のとおりです。
・Thermo Scientific 社製 LCQ、LTQ、LTG-FT、Orbi-Trap
・mzXMLフォーマット(Sashimi Project)など
・その他、詳細はお問合わせ下さい。
【Q】
市販糖鎖標品の分析データを用いて解析を実施してみましたが糖鎖由来データとして検出してくれません。
【A】
測定機種によってはMSMSのスキャンレンジ全域にバックグラウンドピークが立っている場合があります。読み込んだデータのMSMSのクロマトの縦軸を最大(x1000)まで拡大表示させて確認をしてみて下さい。バックグラウンドピークが存在する場合は、「number of peaks」のパラメーター値を10前後の数値ずつ増やして行きながら、条件検討を行ってみて下さい。
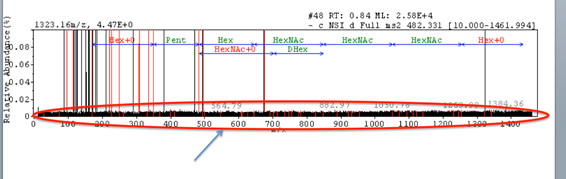
【Q】
解析パラメーターの条件検討をしていますが、毎回Preference => Properties => Functionタブを開いて値を設定しなおしています。もっと簡単な手順で解析パラメーターの検討ができる方法はありませんか?
【A】
画面左のツリー表示のAnalysisの行を右クリックし「Set Default」を選択します。これによりAnalysisに使用したパラメータ値がDefault値として登録されます。
1.「Add Analysis」で解析を実行する前に表示されるウィンドウでパラメーター値を変更
2.解析実行後に結果を確認して、良さそうであれば上記手順でDefault値を変更
3.さらに次のパラメーター値を検討 or 次のデータを解析
上記の手順を繰り返すことで条件検討をスムーズに行って頂けます。
【Q】
マニュアル等に記載の「抽出」と「検出」の違いは何なのでしょうか?
【A】
「抽出ピーク」= Fittingパラメータで解析対象のピークとして抽出されたピーク
「検出ピーク」= Identificationパラメータで糖鎖関連のピークとして検出されたピーク
という意味でマニュアルetcの文章中では使い分けております。
解析結果は保存可能です。
解析パラメータごとに、別名を付けて保存することも可能です。
画像ファイルとしてイメージ出力することができ、論文等にご利用いただくことが可能です。
画像ファイルとしてイメージ出力する場合には、GlyfinTMS のメニューより、[File]->[Export] とお進みください。
イメージ画像のファイルサイズは以下のサイズから選択可能です。
・1680 × 1050
・1600 × 1024
・1280 × 1024
・1024 × 768
・800 × 600
【Q】
「charge」のパラメーターで、「1+」のみチェックを入れた状態で、解析を実行した後にマニュアルでアサインした際、2価や3価の単糖m/z値でもアサインされてしまいます。
【A】
一連のパラメーター値は、自動解析の際にのみ有効な値です。マニュアルアサインでは、単糖と、アミノ酸の登録値を流用しており純粋にドラッグしたピーク間のm/z値が、登録してある何かと一致するかどうかを見ております。(その際に、1価〜3価までのm/z値を含めて見ています)
【Q】
「difference of m/z」は、「range of m/z」と何がちがうのですか?
【A】
以下の図をご覧ください。
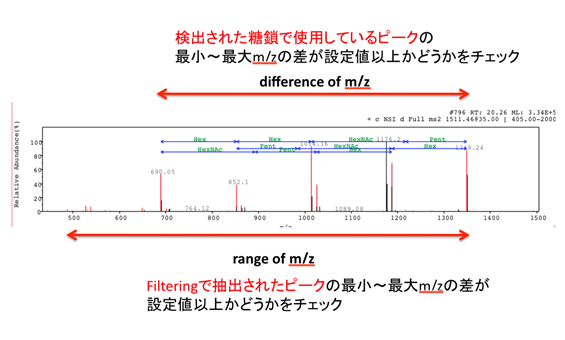
【Q】
MSMSスキャンで、解裂せずに残っているparent ionのピーク強度に対し,fragment peakの強度がかなり低いのですが,この場合解析に何か影響が出てきますでしょうか?
【A】
強度に関するパラメーターの「Intensity」では、スキャン中のBasePeakとの相対強度を見ていますので、parent ionのピーク強度と、fragment peakの強度比は特に影響しないはずです。
【Q】
「Result top」のパラメーターで、「検出された糖鎖を表示する数」と説明がありますが
何か閾値を設けて表示させる順番を決めているのでしょうか?
【A】
抽出ピーク群にRelative Abundance(Intencity)の高い順に値を
付加(順番付け)しておき、検出された1つの糖鎖として使用したピーク群の和が
高いものから順番に「Result top」で指定された数を表示します。より強度の高いピークを使用していて糖鎖と検出されたもの程、表示される順位が高くなります。
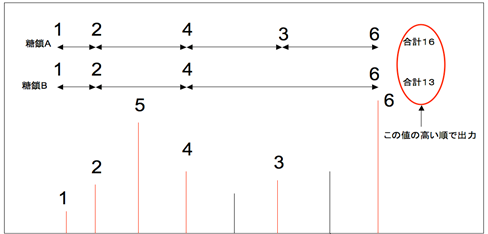 ※赤色ピーク抽出ピーク(糖鎖検出に使用)、黒色ピーク抽出されていない(糖鎖検出に未使用)
※赤色ピーク抽出ピーク(糖鎖検出に使用)、黒色ピーク抽出されていない(糖鎖検出に未使用)
【Q】
Thermo社 LCQで取得したデータを解析したいのですが、パラメーターの設定が合わない様で困っています
【A】
LCQを含む、旧型の出力データを解析する際は以下のパラメータ値をデフォルト値として、この値から少しずつ値を変更して御自分のデータに対してパラメータを最適化して下さい。
| width | → 2.3 |
| maximum candidate peaks | → 2 |
| number of peaks | → 5 < 90 |
| range of m/z | → 645 |
| tolerance | → 1.2 |
| minimum length | → 4 |
| number of identified peaks | → 5 < 45 |
| difference of m/z | → 645 |
| rate of major | → 50 |
| intensity | → 40 |
| charge | → 1+ 2+ |
| maxmum number of minor | → 3 |
| result top | → 5 |